Virologische Analysen im Nationalen Referenzzentrum für Influenzaviren Saison 2023/24
Genetische Charakterisierung der Influenzaviren
Aus Proben des ARE-Praxis-Sentinels (n = 384, blau), des SARI-Krankenhaus-Sentinels (n = 47, rot) sowie aus Proben von anderen Einsendungen (n = 62, lila) wurden 493 Influenzaviren der Saison 2023/24 sequenziert und das Hämagglutinin (HA) genetisch analysiert (Abb. 1). Unter den sequenzierten Influenzaviren gehörten 419 zum Subtyp A(H1N1)pdm09, 33 zum Subtyp A(H3N2) und 41 Viren zur B/Victoria-Linie. Entsprechend ECDC/WHO-Richtlinien wurden die Viren zu Referenzviren und entsprechenden Kladen zugeordnet (Tabelle 1).
BilderstreckeBildVideo
1 / 3

 A(H1N1)pdm09-Viren
A(H1N1)pdm09-Viren
Abb. 1: Phylogenetische Analyse des Hämagglutinin (HA)-Gens von repräsentativen A(H1N1)pdm09-, A(H3N2)- und B/Victoria-Viren aus 2023/24. Die Proben wurden in den Kalenderwochen 05 – 08/2024 entnommen. Die Virusgenome wurden aus Patientenproben vom ARE-Praxis-Sentinel (blau), SARI-Krankenhaus-Sentinel (rot) und von anderen Einsendungen (lila) gewonnen, über Next Generation Sequencing (NGS) sequenziert und die Vollgenome unter www.gisaid.org hinterlegt. Influenzaviren von Patientinnen und Patienten mit einem schweren bis fatalen Verlauf (schwererV), z.B. aufgrund einer Pneumonie und Influenzaviren von geimpften Patientinnen und Patienten (Impfdurchbruch/ImpfD) sind bevorzugt abgebildet. Weiterhin sind Viren mit reduzierter Reaktivität gegenüber dem Impfstoffstamm (reduz. ImpfstammR) dargestellt sowie Viren, die nach über sieben Tagen Erkrankung noch in hohen Konzentrationen in den Patientenproben nachweisbar waren (<ct25/>7T). Darüber hinaus sind ECDC/WHO-Referenzviren und Referenzviren, die für die genetische Zuordnung der zirkulierenden Viren bestimmt wurden, abgebildet. Die abgeleiteten Aminosäuresubstitutionen und Deletionen (-) im HA sind angegeben.
Methode phylogenetische Analyse: Mafft v. 7.520 (Accuracy-based Algorithmus: E-INS-i) für die Erstellung eines Multiplen Sequenz Alignments; Iqtree v. 2.2.2.7 (ML Methode) mit automatischer Selektion des Substitutionsmodels (FLU+I für H0N0, H3N2 und FLU+G4 für H1N1) und 1000 Bootstrap Wiederholungen für die Erstellung der Phylogenie; TreeTime v. 0.11.1 für die Rekonstruktion anzestraler Sequenzen und Annotation der Zweige mit Mutationen.
Tabelle 1: HA-Analyse und Zuordnung der zirkulierenden Influenzaviren in 2023/24 (Probeneingang ab KW40/2023) zu Referenzviren und Kladen (ECDC/WHO)
| Referenzvirus | GISAID Isolat Accession-Nr. | Klade | n |
|---|
| Gesamt | | | | 493 |
A(H1N1pdm09)
| A/Victoria/2570/2019_Egg | EPI_ISL_417210 | 5a.2 | 0 |
| A/Sydney/5/2021_Cell | EPI_ISL_8767089 | 5a.2a | 369 |
| A/Wisconsin/67/2022_Cell | EPI_ISL_15928563 | 5a.2a.1 | 0 |
| A/Victoria/4897/2022_Egg | EPI_ISL_17072386 | 5a.2a.1 | 50 |
| A(H3N2) | A/Darwin/9/2021_Egg | EPI_ISL_3801278 | 2a | 0 |
| A/Catalonia/NSVH161512067/2022 | EPI_ISL_16043979 | 2a.1b | 0 |
| A/Finland/402/2023 | EPI_ISL_18237981 | 2a.3a | 0 |
| A/Thailand/8/2022 | EPI_ISL_16014504 | 2a.3a.1 | 33 |
| A/Sydney/732/2022 | EPI_ISL_16003445 | 2a.3b | 0 |
| A/Thüringen/10/2022 | EPI_ISL_13704107 | 2b | 0 |
| B/Victoria | B/Washington/02/2019_Egg | EPI_ISL_362540 | V1A.3 | 0 |
| B/Austria/1359417/2021_Cell | EPI_ISL_983345 | V1A.3a.2 | 0 |
| B/Moldova/2030521/2023 | EPI_ISL_18037429 | V1A.3a.2 | 0 |
| B/Connecticut/01/2021 | EPI_ISL_13912922 | V1A.3a.2 | 13 |
| B/Catalonia/2279261NS/2023 | EPI_ISL_18109004 | V1A.3a.2 | 28 |
nach oben
Antigenetische Charakterisierung der Influenzaviren
Seit der KW40 2023 wurden 317 Influenza A(H1N1)pdm09-Viren, 51 Influenza A(H3N2)-Viren und 84 Influenza B-Viren der Victoria-Linie in Zellkultur isoliert. 210 A(H1N1)pdm09-Virusisolate, sechs A(H3N2)-Isolate und 49 B/Victoria-Virusisolate wurden von anderen Einrichtungen eingesandt und in die antigene Charakterisierung einbezogen. Die A(H1N1)pdm09-Viren reagierten sehr gut mit dem entsprechenden Referenzserum (Impfstoffvirus A/Victoria/4897/2022; Abb. 2). Da gegenwärtig hauptsächlich A(H1N1)pdm09 Influenzaviren der Clade A/Sydney/5/2021 zirkulieren, wurden die Viren auch gegen dieses Virus getestet. Die Viren reagierten entweder gleich stark oder größtenteils eine log2-Titerstufe höher als mit A/Victoria/4897/2022 (Daten nicht gezeigt). Alle isolierten A(H3N2)-Viren wurden vom gegen den Impfstamm gerichteten Referenzserum (Impfstoffvirus A/Darwin/9/2021) im Hämagglutinationshemmtest gut erkannt, jedoch reagierten die aktuellen Isolate deutlich schwächer als das Impfstoffvirus (Abb. 3). Da A(H3N2) Viren der Klade 2a.3a.1 in den Nachweisen dominierten wurde für die nächste Saison ein Impfstoffvirus dieser Klade empfohlen, das A/Darwin/9/2021 ersetzen wird. Die B/Victoria-Viren wurden vom Impfstammantiserum (B/Austria/1359417/2021) ebenfalls detektiert, reagierten aber größtenteils schwächer als das Impfstammvirus (Abb. 4).
Diese Analysen dienen der Untersuchung der Passgenauigkeit der Impfstämme; sie erlauben keine vollständigen Aussagen zur Wirksamkeit der Impfstoffe, da hier noch andere Faktoren berücksichtigt werden müssen (Abstand zur letzten Impfung, Zahl vorangegangener Antigenkontakte, Expositionsdosis, Alter u.a.).

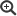
Abb. 2: Reaktivität isolierter A(H1N1)pdm09-Influenzaviren mit gegen den Impfstamm A/Victoria/4897/2022 gerichtetem Frettchenimmunserum. Rot, Geometrisches Mittel der Positivkontrollen (homologe Reaktion gegen das Impfstammvirus); blau, Reaktivität der Isolate; grün Bereich von 2 log2 um den Mittelwert der Positivkontrollen = Bereich optimaler Passgenauigkeit in Bezug zum Impfstamm. Quelle: RKI

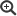
Abb. 3: Reaktivität isolierter A(H3N2)-Influenzaviren mit gegen den Impfstamm A/Darwin/9/2021 gerichtetem Frettchenimmunserum. Rot, Geometrisches Mittel der Positivkontrollen (homologe Reaktion gegen den Impfstamm); blau, Reaktivität der Isolate; grün Bereich von 2 log2 um den Mittelwert der Positivkontrollen = Bereich optimaler Passgenauigkeit in Bezug zum Impfstamm. Quelle: RKI

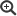
Abb. 4: Reaktivität isolierter B/Victoria-Influenzaviren mit gegen den Impfstamm B/Austria/1359417/2021 gerichtetem Frettchenimmunserum. Rot, Geometrisches Mittel der Positivkontrollen (homologe Reaktion gegen den Impfstamm); blau, Reaktivität der Isolate; grün Bereich von 2 log2 um den Mittelwert der Positivkontrollen = Bereich optimaler Passgenauigkeit in Bezug zum Impfstamm. Quelle: RKI
nach oben
Resistenzanalyse der Influenzaviren
Es wurden keine Resistenzen gegen Neuraminidaseinhibitoren (Oseltamivir, Zanamivir) bei den bisher untersuchten Viren nachgewiesen; hierbei wurden 137 A(H1N1)pdm09-, 33 A(H3N2)-Viren und 22 B/Victoria-Viren untersucht.
nach oben
Zirkulation respiratorischer Viren
Gegenwärtig dominieren endemische Coronaviren (überwiegend HKU1 und OC43), Rhinoviren, Influenzaviren (hauptsächlich B/Victoria) und Parainfluenzaviren (größtenteils PIV-3) das Infektionsgeschehen, aber auch andere respiratorische Viren werden detektiert. Bei den Influenzaviren gehen die Nachweise von A(H1N1)pdm09 zurück und die der B/Victoria-Viren steigen an (Abb. 5). Die Positivenraten von RSV fallen und die RSV-Welle endete in KW10 2024 (Abb. 6). SARS-CoV-2 wurden von KW34-52 2023 regelmäßig mit Positivenraten >15% im ARE-Praxis-Sentinel nachgewiesen; seit KW1 2024 gingen die Positivenraten zurück und bewegten sich ab Anfang Februar 2024 auf dem Niveau der endemischen Coronaviren; in KW13 2024 gab es keinen Nachweis von SARS-CoV-2 im Sentinel (Abb. 7).

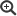
Abb. 5: Zirkulation von Influenzaviren von der letzten präpandemischen Saison bis jetzt (ARE-Praxis-Sentinel). Quelle: RKI

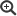
Abb. 6: Zirkulation von Rhinoviren (HRV), Parainfluenzaviren (PIV), Humanen Metapneumoviren (HMPV) und Respiratorischen Synzytialviren (RSV) von der letzten präpandemischen Saison bis jetzt (ARE-Praxis-Sentinel). Quelle: RKI

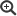
Abb. 7: Zirkulation von SARS-CoV-2 und endemischen Coronaviren (229E, HKU1, OC43, NL63) von der letzten präpandemischen Saison bis jetzt (ARE-Praxis-Sentinel). Quelle: RKI
nach oben
Surveillance schwere akuter respiratorischer Infektionen
Neben dem virologischen Sentinel im ambulanten Bereich werden auch Proben von stationär aufgenommenen Patienten untersucht. Seit der KW40 2023 wurden hier vor allem SARS-CoV-2, RSV und A(H1N1)pdm09 Influenzaviren nachgewiesen. Die Nachweise von A(H1N1)pdm09-Influenzaviren im stationären Bereich waren bis zur KW5 2024 deutlich angestiegen und gehen seitdem zurück; in KW13 2024 gab es keinen Nachweis (Abb. 8). Die RSV-Nachweise haben nach einem Peak in KW1 2024 abgenommen, sich über zwei Monate auf einem Niveau mit Positivraten zwischen 10 und 20% bewegt und werden gegenwärtig nicht mehr so häufig im stationären Bereich detektiert (Abb. 9). Die SARS-CoV-2-Nachweise in Krankenhäusern sind seit Anfang des Jahres 2024 rückläufig und in den letzten beiden Wochen wurden keine SARS-CoV-2 im stationären Sentinel nachgewiesen (Abb. 10).

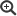
Abb. 8: Nachweis von Influenzaviren von KW40 2023 bis jetzt (SARI-Sentinel). Quelle: RKI

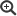
Abb. 9: Nachweise von Rhinoviren (HRV), Parainfluenzaviren (PIV), Humanen Metapneumoviren (HMPV) und Respiratorischen Synzytialviren (RSV) von KW40 2023 bis jetzt (SARI-Sentinel). Quelle: RKI

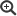
Abb. 10: Nachweise von SARS-CoV-2 und endemischen Coronaviren (229E, HKU1, OC43, NL63) von KW40 2023 bis jetzt (SARI-Sentinel). Quelle: RKI
nach oben